 位置:
学术进展
位置:
学术进展2023年10月13日,庞尔丽教授课题组在Briefings in Bioinformatics(五年影响因子10.6)发表题为“MkcDBGAS: a reference-free approach to identify comprehensive alternative splicing events in a transcriptome”的研究论文,提出了在无参考基因组的情况下,识别可变剪切的方法。
可变剪切(alternative splicing)作为一种重要的转录后修饰机制,调节许多生物过程,同时是表型多样性的重要来源,对生物的环境适应和进化发挥着重要作用。然而,在没有参考基因组的指导下,识别可变剪切事件仍然是一个挑战。
本研究提出了一种新的方法—MkcDBGAS,在没有参考基因组的情况下,只利用转录组来识别所有七种类型的可变剪切事件。MkcDBGAS由三个模块组成(图1),分别利用人类和拟南芥的全长转录本构建了两个模型。在第一个模块中,MkcDBGAS首次使用混合kmers的着色de Bruijn 图来识别由于可变剪切而产生的气泡(bubbles),在人类和拟南芥的转录组中,准确度分别为98.17%和99.31%;召回率为93.45%和95.34%;并且可以识别其它方法所不能识别的可变剪切事件。在第二个模块中, MkcDBGAS首次使用外显子的基序(motifs)构建了特征矩阵,并基于XGBoost的分类器对可变剪切事件进行分类,极大的提升了分类器的正确率,在人类和拟南芥的模型中正确率分别达到93.44%和94.09%;并且优于其它的机器学习模型和已有的分类方法(图2)。在第三个模块中,当转录本表达量数据可以获得时,MkcDBGAS提供了在多种条件下差异可变剪切的分析。把MkcDBGAS应用于人类的两个细胞系(PC3E和GS689)、老鼠的转录组和无油樟(Amborella)的三代测序(isoform sequencing,Iso-Seq)转录组4个真实数据集中,可变剪切转录本识别的准确度都达到98.37%以上,召回率高于93.02%,分类正确率也都高于90.96%,说明MkcDBGAS是高度可扩展的,可以应用于其他物种中。MkcDBGAS首次仅利用转录组数据来识别所有类型的可变剪切事件,这将极大地促进可变剪切在更广泛领域的研究。
北京师范大学生命科学学院生态系已毕业博士研究生张泉宝为论文第一作者,已毕业硕士研究生曹磊、宋宏涛博士和林魁教授也参与了研究。庞尔丽教授为论文的通讯作者,抗性基因资源与分子发育北京市重点实验室为第二单位。该研究得到了国家自然科学基金和中央高校基本科研业务费专项资金的资助。
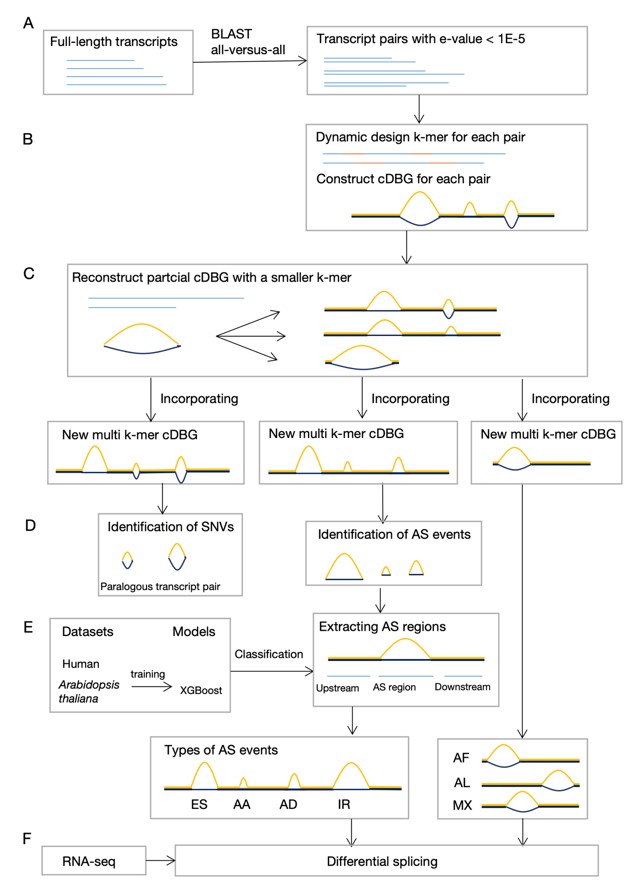
图1 MkcDBGAS的工作流程。A-D)混合kmers着色de Bruijn 图识别可变剪切转录本。E)基于XGBoost可变剪切事件的分类器。F)差异可变剪切分析。
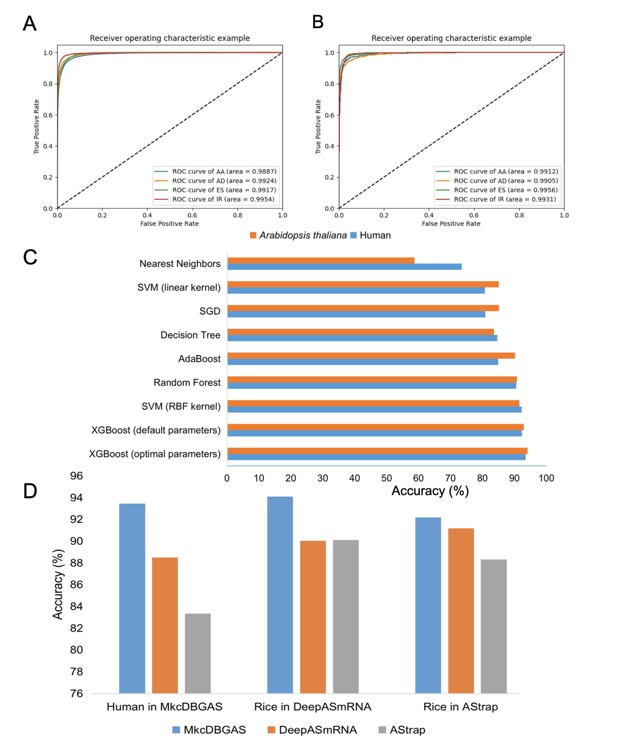
图2 MkcDBGAS分类器表现
原文链接:https://academic.oup.com/bib/article/24/6/bbad367/7313457