 位置:
学术进展
位置:
学术进展2023年6月24日,韩生成与张凡凡老师课题组在International Journal of Molecular Sciences(IF:6.208)发表题为“Transcriptome screening of long non-coding RNAs and their target protein coding genes unmasks a dynamics portrait of seed coat coloration associated with anthocyanins in Tibetan hulless barley”的研究性文章。抗性基因资源与分子发育北京市重点实验室直博生郑凯峰为第一作者,韩生成与张凡凡老师为共同通讯作者。
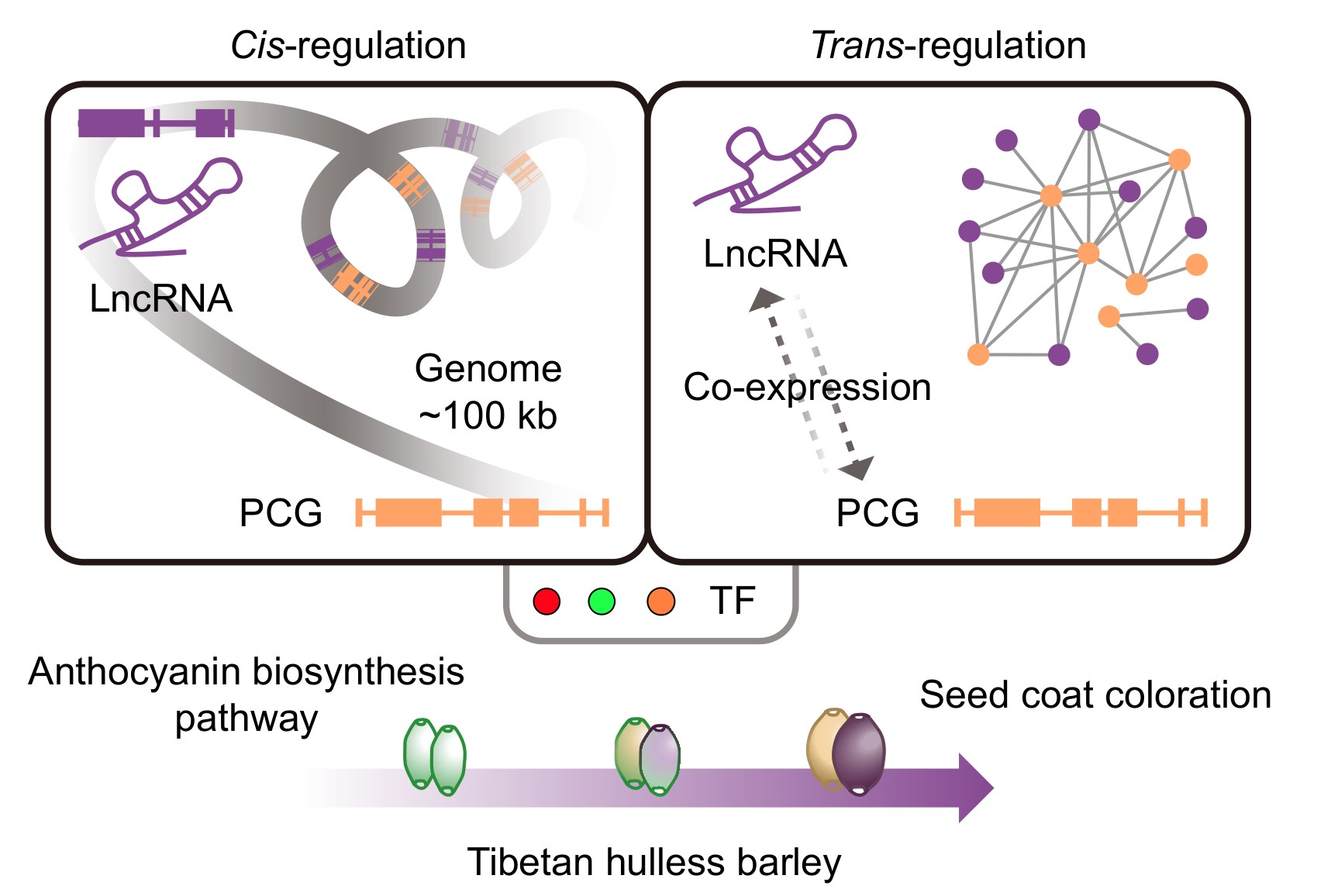
青稞俗称裸大麦,是青藏高原地区的口粮作物,也是我国最重要的杂粮作物之一。青稞具有“四高四低”(高蛋白、高可溶性纤维、高维生素、高矿物质;低脂肪、低糖、低胆固醇、低钠)的营养成分构成,也是最重要的健康食品。许多植物都拥有积累花青素并利用其着色的能力,而花青素对人类健康也非常有益。在青稞中,目前对花青素形成机制的研究仅仅停留在揭示其合成代谢的蛋白编码基因(PCG)的水平。本项研究中,我们基于最新公布的青稞基因组,对已发表的紫色和白色青稞种皮中沿发育梯度的转录组数据进行了深入的生物信息学挖掘,共鉴定出9414个长链非编码RNAs(大于200 nt, lncRNAs)。首先,在全转录组范围记录了青稞lncRNAs的特征:GC含量波动、长度不均匀、外显子数量类型和转录本分类多样。另外,还发现了一些青稞lncRNAs在大麦和其他单子叶植物中存在可检测的序列保守性。进一步对白青稞和紫青稞中具有显著差异表达的lncRNAs(DElncRNAs)和PCGs(DEPCGs)进行分析。结果表明,这些差异表达基因主要集中在种子发育的后期。对lncRNA调控靶基因的作用分析表明,DElncRNAs一方面可能顺式调控与多种花青素合成途径相关的DEPCGs,包括乳熟末期和蜡熟初期的黄酮类和花青素生物合成。另一方面,基因共表达分析结果也表明,lncRNAs可能通过反式调节上调花青素合成途径上的PCGs进而影响种皮颜色。另外,青稞lncRNAs和许多转录因子的交织也可能在种皮着色中发挥作用。因此,通过本项目的研究,首次绘制了包含青稞lncRNAs的花青素合成途径动态图谱。该工作扩充了选育健康营养的新型“紫色谷物”的遗传资源库,并从lncRNA的角度提供了更多关于青稞花青素合成的见解,有助于后续作物分子设计育种的发展。
该研究受国家自然科学基金(32270346)、青海省科学技术厅(2023-ZJ-706)与青海省“昆仑英才·高端创新创业人才”计划(QHCH-2022-027)等的资助。
全文链接:https://www.mdpi.com/1422-0067/24/13/10587